Polimorfizm pojedynczego nukleotydu
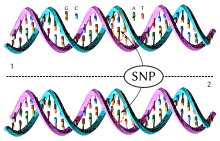
Polimorfizm pojedynczego nukleotydu (ang. Single Nucleotide Polymorphism w skrócie SNP) – zjawisko zmienności sekwencji DNA, która polega na zmianie pojedynczego nukleotydu (A, T, C lub G) pomiędzy osobnikami danego gatunku lub drugim, odpowiadającym chromosomem danego osobnika.
Przykładowo w dwóch sekwencjach DNA od różnych osobników, AAGCCTA i AAGCTTA, występuje różnica w jednym nukleotydzie. W tym wypadku mówimy o 2 allelach: C i T.
SNP stanowią ok. 90% całej zmienności występującej w ludzkim genomie i występują co 100-300 nukleotydów na ogólną ich liczbę 3 mld.
W badaniach populacyjnych można ustalić względną częstość występowania danego wariantu związanego z SNP. Należy stwierdzić, że występują różnice w występowaniu SNP pomiędzy różnymi geograficznie lub etnicznie populacjami (np. w genie SLC24A5 i kolorze skóry).
Stwierdzane w materiale genetycznym przypadki polimorfizmu pojedynczego nukleotydu mogą być stwierdzane w sekwencjach kodujących genów, regionach niekodujących genów lub w regionach międzygenowych. Polimorfizm pojedynczego nukleotydu w sekwencji kodującej jakiegoś genu nie musi koniecznie prowadzić do zmiany w sekwencji aminokwasowej białka ze względu na degenerację kodu genetycznego. SNP dzieli się z tego względu na synonimiczne lub niesynonimiczne (kiedy sekwencja powstałego białka ma zmieniony aminokwas w związku z polimorfizmem). Przypadki polimorfizmu pojedynczego nukleotydu, które stwierdza się poza sekwencjami kodującymi genów, mimo to mogą wywierać działania biologiczne (np. zmiana w sekwencji RNA lub w sekwencji regulującej ekspresję genu).
Zmiany w sekwencji ludzkiego DNA mogą wywoływać choroby, zmieniać odpowiedź organizmu na pewne patogeny, substancje chemiczne, leki itp. Jednak największe znaczenie mają badania porównawcze DNA osób chorujących na dane schorzenie i bez tej choroby (marker genetyczny).
Zobacz też
edytujLinki zewnętrzne
edytuj- Informacje o SNP w Human Genome Project
- International HapMap Project. hapmap.org. [zarchiwizowane z tego adresu (2014-04-16)].
- 1000 Genomes Project